# Configuração inicial e importação de bibliotecas
import pymc as pm
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
from scipy import stats
# Configurações para plots
plt.style.use('seaborn-v0_8-darkgrid')
plt.rcParams['figure.figsize'] = (9, 6)Modelo Normal Bayesiano
Modelando Dados Contínuos no PyMC - distribuições a priori
Exploraremos a inferência bayesiana, com foco na modelagem de dados contínuos por meio da distribuição normal. Nosso objetivo será desenvolver a intuição sobre como escolher distribuições a priori e como o PyMC nos auxilia a visualizar as consequências dessas escolhas sobre a distribuição preditiva a priori da variável de interesse. Para isso, utilizaremos um exemplo baseado na distribuição de altura em adultos.
- Apresentar e compreender a distribuição normal de forma intuitiva.
- Simular dados a partir de distribuições normais utilizando
scipy. - Utilizar seu conhecimento subjetivo para propor e visualizar distribuições a priori para os parâmetros de um modelo normal (por exemplo, altura de adultos).
- Visualizar a distribuição preditiva a priori da variável resposta com o PyMC.
1 Explorando a Distribuição Normal
A Distribuição Normal, frequentemente chamada de curva de sino ou curva Gaussiana, é central em estatística. Ela é caracterizada por dois parâmetros: a média (\(\mu\)) e o desvio padrão (\(\sigma\)). A média determina o centro da distribuição, enquanto o desvio padrão determina sua dispersão ou largura. Muitos fenômenos naturais podem ser adequadamente descritos por essa distribuição.
A ideia intuitiva: Pense na altura de adultos. Há um valor central (a média) em torno do qual a maioria das alturas se agrupa. Há também uma variação: algumas pessoas são mais altas, outras mais baixas. O desvio padrão nos diz o quão espalhadas essas alturas tendem a ser em relação à média.
1.1 Curva de densidade de probabilidade
Gerando a Distribuição de Densidade Normal para diferentes valores de \(\mu\) e \(\sigma\).
# Parâmetros da distribuição
mu_1 = 20
sigma_1 = 3
mu_2 = 20
sigma_2 = 6
mu_3 = 30
sigma_3 = 5
# Limites gráficos
x_min = np.min(np.array([mu_1, mu_2, mu_3]) - 4*np.array([sigma_1, sigma_2, sigma_3]))
x_max = np.max(np.array([mu_1, mu_2, mu_3]) + 4*np.array([sigma_1, sigma_2, sigma_3]))
x = np.linspace(x_min, x_max, 1000) # Faixa de alturas para plotar
pdf_1 = stats.norm.pdf(x, loc=mu_1, scale=sigma_1)
pdf_2 = stats.norm.pdf(x, loc=mu_2, scale=sigma_2)
pdf_3 = stats.norm.pdf(x, loc=mu_3, scale=sigma_3)Visualizando as distribuições de densidade de probabilidade
plt.plot(x, pdf_1, label=f'$\mu={mu_1}, \sigma={sigma_1}$', color='b', lw=2)
plt.plot(x, pdf_2, label=f'$\mu={mu_2}, \sigma={sigma_2}$', color='r', lw=2)
plt.plot(x, pdf_3, label=f'$\mu={mu_3}, \sigma={sigma_3}$', color='g', lw=2)
plt.xlabel('X', fontsize=12)
plt.ylabel('Densidade de Probabilidade', fontsize=12)
plt.legend()
plt.grid(True, linestyle='-', alpha=0.7)
plt.show()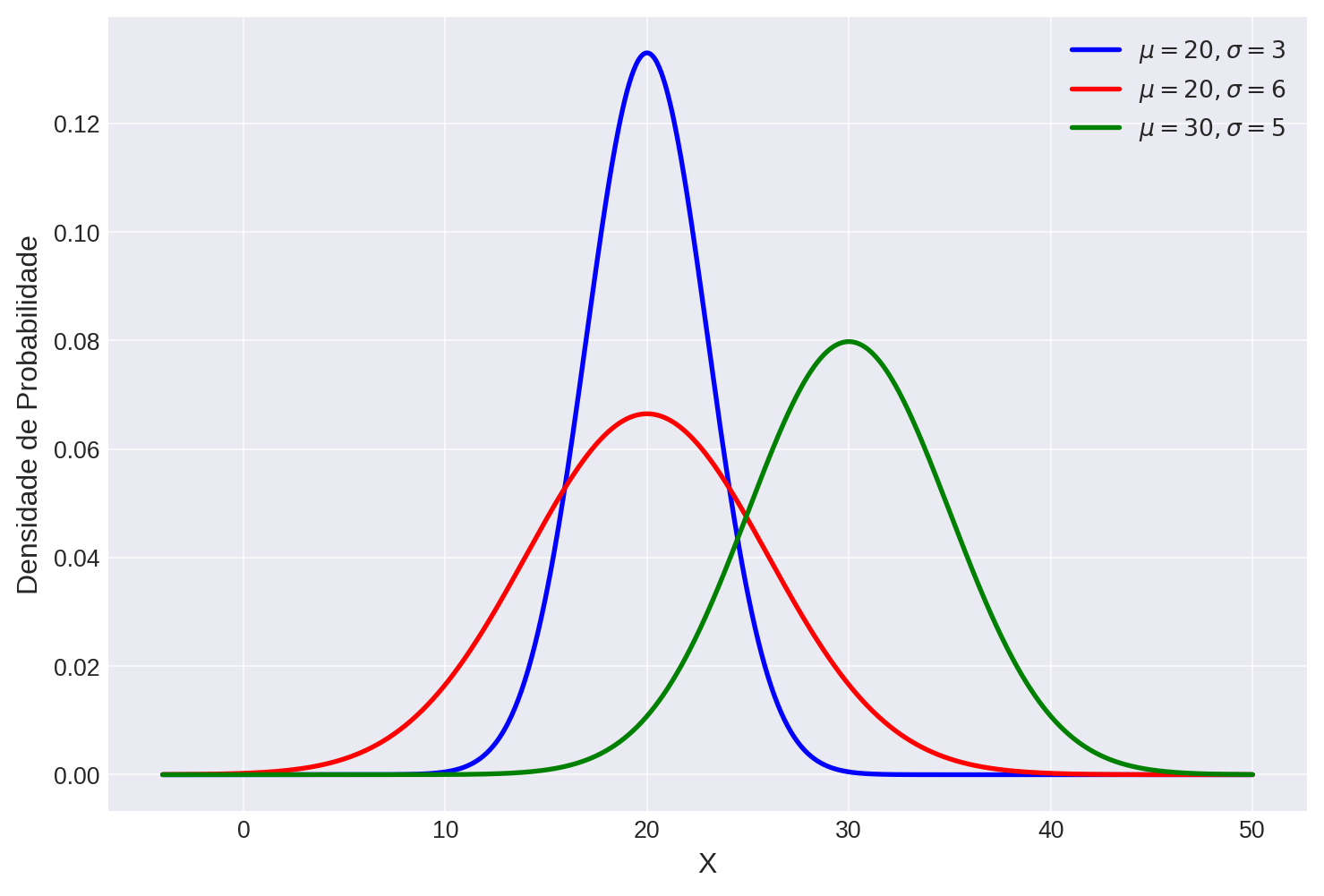
1.2 Amostrando valores de distribuição normal
Na Figura 1 vemos as curvas teóricas de densidade da distribuiçao normal. Podemos também gerar amostras valores ao acaso destas distribuições para verificar como estas amostras se parecem. Isso simula o processo de sortear alturas de uma população que segue essa distribuição.
mu = 20
sigma = 4
num_amostras = 60Verificando o histograma dos valores sorteados.
amostras_y = stats.norm.rvs(loc=mu, scale=sigma, size=num_amostras)
x_dens = np.linspace(mu-4*sigma, mu+4*sigma, 500)
plt.hist(amostras_y, bins=30, density=True, alpha=0.8, color='lightblue', label='Amostras Geradas')
sns.kdeplot(amostras_y, color='blue', linewidth=2, label='Densidade Empírica')
plt.plot(x_dens, stats.norm.pdf(x_dens, loc=mu, scale=sigma), color='red', linewidth=2.5, label='Densidade Teórica')
plt.xlabel('X', fontsize=12)
plt.ylabel('Densidade / Frequência Normalizada', fontsize=12)
plt.legend()
plt.grid(True, linestyle='--', alpha=0.7)
plt.show()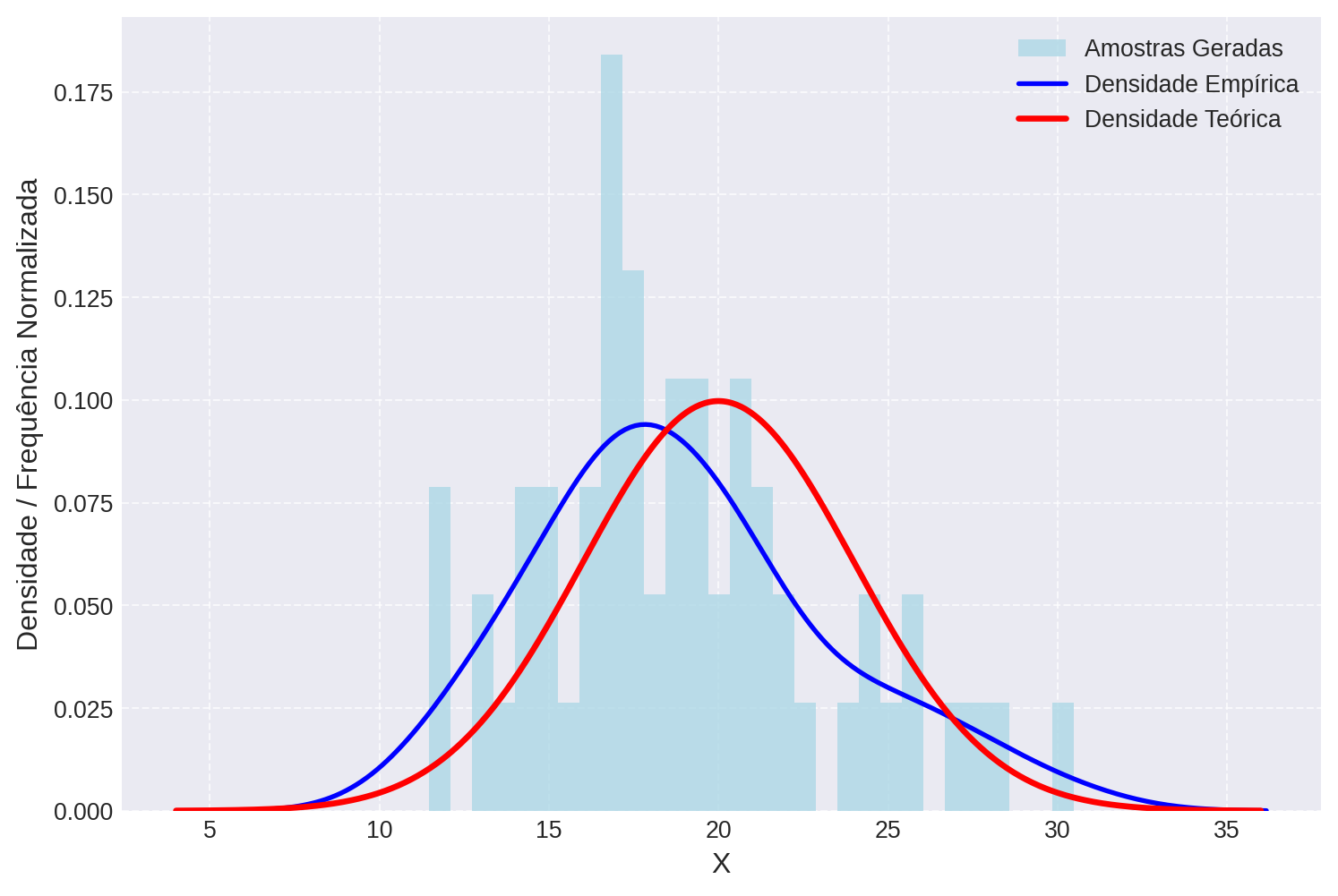
- Rode este o trecho de código acima algumas vezes e observe como se dá a variação amostral.
- Aumente e diminua o tamanho da amostras e verifique a variação entre as curvas enpíricas e a curva teórica.
2 Intuição Bayesiana
Em inferência Bayesiana, começamos com crenças sobre os parâmetros (a priori) e as atualizamos com dados (a posteriori). Para a altura humana (\(y\)), podemos assumir que a distribuição normal é um bom modelo preditivo.
Deste modo, escrevemos que:
\[y \sim \mathcal{N}(\mu, \sigma) \tag{1}\]
Em seguida, precisamos sugerir uma dristribuição a priori para a média \(\mu\) e o desvio padrão \(\sigma\) que traduzam de forma adequada o que esperamos sobre a distribuição de altura em adultos. Sabemos por exemplo que a média da altura de adultos não é 50 cm nem 300 cm. Qual sua intuição sobre o desvio padrão?
- Assumindo que a distribuição de alturas em adultos segue uma dsitribuição normal proponha valores razoáveis para a média (\(\mu\)) e o desvio padrão (\(\sigma\)).
- Para ajudar a decidir sobre o que seriam valores valores razoáveis, plote as curvas de densidade de probabilidade resultante de sua escolha e faça algumas simulações para verificar quais valores estremos sua escolha é capaz de gerar, utilizando os códigos da Seção 1.2.
2.1 Checagem Priori Preditiva
Assumindo que a altura de adultos segue uma distribuição (Equação 1), vamos assumir que o parâmetro \(\mu\) segue também uma distribuição normal e que \(\sigma\) segue uma distribuição log-normal
Como utilizamos estes pressupostos para escolher distribuição razoiáveis para \(\mu\) e \(\sigma\)?
Priori para \(\mu\)
mean_prior_mu = # INSIRA SUA ESCOLHA PARA A MÉDIA DA PRIORI DE mu
sd_prior_mu = # INSIRA SUA ESCOLHA PARA O DESVIO PADRÃO DA PRIORI DE mu
# Gere sequancia de x e calcule a PDF
xmean_prior = np.linspace(mean_prior_mu - 4*sd_prior_mu, mean_prior_mu + 4*sd_prior_mu, 1000)
pdf_mean_prior = stats.norm.pdf(x = xmean_prior, loc = mean_prior_mu, scale = sd_prior_mu)
# Plote os resultados
plt.plot(xmean_prior, pdf_mean_prior)
plt.title(f'Priori para $\mu$')
plt.show()Priori para \(\sigma\)
lmean_prior_sigma = # INSIRA SUA ESCOLHA PARA A MÉDIA DA PRIORI DE sigma
lsd_prior_sigma = # INSIRA SUA ESCOLHA PARA O DESVIO PADRÃO DA PRIORI DE sigma
xsd_prior = np.linspace(0.01, 20, 1000)
pdf_sd_prior = stats.lognorm.pdf(xsd_prior, s=lsd_prior_sigma, scale=lmean_prior_sigma)
plt.close()
plt.plot(xsd_prior, pdf_sd_prior)
plt.title(f'Priori para $\sigma$')
plt.show()Extraindo distribuição a priori preditiva de \(y\) no PyMC
# Definindo o modelo APENAS com as priores compartilhadas
with pm.Model() as prior_predictive_model:
# Priori para a média
mu = pm.Normal("mu", mu = mean_prior_mu, sigma = sd_prior_mu)
# Priori lognormal para o desvio padrão
sigma = pm.Lognormal("sigma", mu = np.log(lmean_prior_sigma), sigma = lsd_prior_sigma)
# Distribuição preditiva de y
y_pred = pm.Normal("y_pred", mu = mu, sigma = sigma)
# Amostras da priori preditiva
prior_predictive_samples = pm.sample_prior_predictive(samples=1000)Agora, vamos visualizar a distribuição dessas amostras preditivas a priori:
y_pred_prior = prior_predictive_samples.prior["y_pred"].values.flatten()
plt.figure(figsize=(10, 6))
plt.hist(y_pred_prior, color='skyblue', edgecolor='black')
plt.xlabel('Alturas priori simulada (cm)', fontsize=12)
plt.ylabel('Densidade', fontsize=12)
plt.grid(True, linestyle='--', alpha=0.7)
plt.show()Olhe para o histograma. As alturas simuladas parecem razoáveis para alturas de adultos? A distribuição faz sentido dada a sua intuição? Se sim, suas priores iniciais eram sensatas. Se não, é importante considerar um ajuste de suas priores (ex: tornar a priori de \(\sigma\) mais restrita se a dispersão for muito grande, ou ajustar a localização/escala da priori de \(\mu\)).
Checagem priori preditiva com PyMC
Podemos chegar não somente a distribuição preditiva de \(y\), mas também dos parâmetros \(\mu\) e \(\sigma\) usando o PyMC. Além disso, poderíamos testar outras distribuições a priori para algum dos parâmetros, por exemplo sigma. Teste cada uma destas e verifique os efeitos sobre as distribuições preditivas.
# Definindo o modelo APENAS com as priores compartilhadas
with pm.Model() as prior_predictive_model:
# Priori para a média
mu = pm.Normal("mu", mu=175, sigma=10)
# Escolha uma das prioris para sigma:
sigma = pm.Lognormal("sigma", mu=np.log(0.08), sigma=0.5)
# sigma = pm.InverseGamma("sigma", alpha=8, beta=0.9)
# sigma = pm.HalfNormal("sigma", sigma=0.1)
# sigma = pm.HalfCauchy("sigma", beta=0.1)
# sigma = pm.Exponential("sigma", lam=20)
# sigma = pm.TruncatedNormal("sigma", mu=0.08, sigma=0.02, lower=0)
# sigma = pm.Uniform("sigma", lower=0, upper=1)
# Distribuição preditiva de y
y_pred = pm.Normal("y_pred", mu=mu, sigma=sigma)
# Amostras da priori preditiva
prior_predictive_samples = pm.sample_prior_predictive(samples=1000)mu_pred_prior = prior_predictive_samples.prior["mu"].values.flatten()
sigma_pred_prior = prior_predictive_samples.prior["sigma"].values.flatten()
y_pred_prior = prior_predictive_samples.prior["y_pred"].values.flatten()
fig, axes = plt.subplots(1, 3, figsize=(9, 3))
axes[0].hist(mu_pred_prior, bins=30, color='skyblue', edgecolor='black')
axes[0].set_xlabel("μ")
axes[0].set_ylabel("Frequência")
axes[1].hist(sigma_pred_prior, bins=30, color='lightgreen', edgecolor='black')
axes[1].set_xlabel("σ")
axes[1].set_ylabel("Frequência")
axes[2].hist(y_pred_prior, bins=30, color='salmon', edgecolor='black')
axes[2].set_xlabel("alturas (y)")
axes[2].set_ylabel("Frequência")
plt.tight_layout()
plt.show()